JWordのO.Yです。
ビッグデータがムーブメントとなって久しく、用途はなんであれ今はHadoopを導入している企業さんも多いことかと思います。
JWordでもHadoopを導入しており検索クエリーの集計等に使用しています。
Hadoopは簡単に分散処理環境を実現することができますが、分散させる処理単位でmapreduceアプリケーションを書かなきゃならないので複雑な処理だとプログラム量が多くなりやすい、Hadoop Streamingを使用した場合には処理スピードが遅い(JWordでは開発効率を優先してPHPかPythonでmapreduceアプリケーションを書いている)、起動デーモンが多すぎる、メモリコントールが難しい(メモリ系の設定がたくさんありバージョンによって微妙に名前が変わってたり廃止されてたりする)、HDFSを必要としない場合でもセットで付いてくる等デメリットもあります。
まあそもそもHadoopはインストール・設定するだけで結構な手間がかかります。CDHを使用したとしてもかなり手間です。
もっといい分散処理基盤はないかな・・・ということで登場したのがApache Sparkです。
ということで今回はApache Sparkに触れてみたいと思います。
(私は脳内では親しみをこめてApacheスパー子と呼んでいます。)
Apache SparkはNext Hadoopということで開発された分散処理基盤です。
Hadoopと比較すると主に以下のような利点があります。(但し、Hadoop抜きで使用する場合のものもあります。)
・構成が単純でわかりやすく、環境構築が容易。おそらく管理もHadoopに比べたら楽だと思われる
・scalaで書ける
・mapperとreducerと分けてプログラムを書く必要がなく、すべての処理を一つにまとめられる
・分散環境をあまり意識せず分散処理を書ける
・Apache Spark専用の機械学習用ライブラリが元々用意されている(Spark MLlib)
・処理データを直接メモリから読んだり書いたりするので処理スピードが速い
・Linuxファイルシステムから直接ファイルの読み込み、書き込みが可能
・mongo-hadoopコネクターを使用することによりMongoDBに対するデータの読み書きが可能
等です。
具体的事例としてwikipediaのページデータをMongoDBから読み込んでSpark MLlibが提供する1機能であるk-meansクラスタリング機能でwikipediaのページをクラスタリングしてみたいと思います。
(但し今回はあくまでApache Sparkの紹介なのでMongoDBの詳細は割愛させていただきます。またブログ掲載のプログラムのインストールは、自己責任で御願いします。インストール等の結果にかかるハードウェアの不稼働等は、 当ブログでは一切サポートしておりませんので、予め御了承下さい。)
使用したApache Spark・MongoDBのバージョンとサーバー環境は以下の通りです。
Apache Spark:1.5.1(Pre-build for Hadoop2.6 and later)
MongoDB:3.0.5
サーバー環境:Conoha(5core メモリ8G) x 5
(ちなみにGMOグループには「サバろうぜ!」というエンジニア支援制度があり、これを利用すると無料で3か月グループ内のサーバーリソースをレンタルすることができます。)
0.下準備・前提
・すべてのサーバー上でsparkユーザーを作成してください。sparkユーザーがApache Spark,Sparkアプリケーションの実行ユーザーとなります。
・今回はApache Sparkのスタンドアローンクラスタ環境を構築します。マスターノード及びワーカーノード起動の際にApache Sparkはマスターノードから各ワーカーノードへ実行ユーザー(spark)でssh通信を実施しますので予めマスターノードから各ワーカーノード及びマスターノード自身にsparkユーザーで公開鍵認証でログインできるように設定をしておいてください。
・スタンドアローンクラスタのマスター・ワーカー起動時、Sparkアプリケーション実行時に関係するプロセスが複数ポートでサーバー間通信を行います。ですので予め使用されるポートを開けておく必要があります。今回は同一ネットワークセグメントのサーバーからの通信に対しては全ポートを開けています。
・rubyとpythonを使用しますのでこちらもインストールしておいてください。バージョンはrubyは最新、pythonは2.7でいいです。
rubyはwp2txtを使用してwikipedia記事のXMLベースのアーカイブファイルをテキストファイルに変換するために、pythonはMongoDBにwikipediaのデータを登録するのに使用します。pythonでMongoDBにアクセスするためににサードパーティーライブラリのpymongoが必要になりますのでpipを使用してインストールしておいてください。
wp2txtのインストールについてはwp2txtでwikipediaのコーパスを作るまでの道のりを参考にしてください。
wikipedia記事データの最新アーカイブは以下のリンクからjawiki-latest-pages-articles.xml.bz2を選択してダウンロードしておいてください。
wikipedia最新各種アーカイブページ
・Apache Sparkはscalaで開発されているため当然scala本体とscalaで書いたユーザーアプリケーションのコンパイルにOpenJDKが必要になりますのでこちらもあらかじめインストールしておいてください。scala本体はhttp://www.scala-lang.org/download/all.htmlからダウンロード、インストールしてください。OpenJDKはyumでインストールが可能です。
今回使用したscalaは2.10.5,OpenJDKは1.8.0_65となります。
・各サーバーのホスト名をあらかじめ設定しておいてください。今回は5台の内1台をマスターノード、4台をワーカーノードとして割り当て、ホスト名はマスターノードはmaster,各ワーカーノードはnode1~node4にしてあります。また全サーバーの/etc/hostsに前述したホスト名とIPの対応関係を記述しておいてください。
・MongoDBはhttps://www.mongodb.org/downloads#productionから使用するサーバーに合うバージョン3系のディストリビュージョンをダウンロード、インストールしてください。
また今回使用したMongoDBの環境はmongos1台、mongoconfig1台(mongosと同居),プライマリ、セカンダリそれぞれ2台ずつの5台環境(Apache Sparkのマスターノード、ワーカーノードと同じサーバーにインストール・起動してあります)となっておりかつwikipediaのアーカイブデータをシャーディングにより分散配置しました。しかし今回紹介する事例に使用したアプリケーションはスタンドアローン環境、単一のプライマリ、セカンダリ構成でも動作します。
・最新バージョンのmecab、mecab用辞書(ipaidc)をインストールしておいてください。これはwikipediaの各ページデータの形態素解析に使用します。
最新バージョンのmecab本体及びipadicはMeCab: Yet Another Part-of-Speech and Morphological Analyzerからダウンロードできます。
・最新バージョンのmecab-Javaバインディングのダウンロードとビルド
上記mecabのダウンロードページと同じページにmecabをJavaで使用するためのバインディングがあるのでそれをダウンロードしてビルドしておいてください。
ビルド後にMeCab.jarとlibMeCab.soが生成されるのでMeCab.jarは後述するsbtのライブラリディレクトリに、libMeCab.soは/usr/local/libに移動してください。
・Apache Sparkの基本的な知識に関しては今回は割愛させていただきますので他の参考ページをご参照ください。
1.Apache Sparkの入手とインストール・スタンドアローンクラスター起動
Apache Sparkのソースとビルド済みバイナリは以下のページで配布されています。
http://spark.apache.org/downloads.html
現在の最新バージョンは1.5.2ですが、ダウンロード当時は1.5.1が最新で、今回も1.5.1を使用してます。
上記ページで「1.Choose a Spark release:」として1.5.1を、「2.Choose a package type:」としてPre build for Hadoop2.6 and laterを選択します。
そうすると「4.Download Spark:」の右横に選択した条件を満たすリソースのダウンロードが可能なミラーサーバーの一覧ページのリンクが表示されますのでクリックし遷移します。後は適当なミラーサーバーからspark-1.5.1-bin-hadoop2.6.tgzをマスターノード側からwgetを使用してダウンロードして解凍します。
|
1 2 3 |
wget http://ftp.jaist.ac.jp/pub/apache/spark/spark-1.5.1/spark-1.5.1-bin-hadoop2.6.tgz tar xfvz spark-1.5.1-bin-hadoop2.6.tgz cd spark-1.5.1-bin-hadoop2.6 |
解凍して生成されたディレクトリの中身は以下のように感じになっています。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 |
drwxr-xr-x 3 spark spark 4096 Nov 25 17:24 bin -rw-r--r-- 1 spark spark 960539 Sep 24 15:13 CHANGES.txt drwxr-xr-x 2 spark spark 4096 Nov 25 13:02 conf drwxr-xr-x 3 spark spark 4096 Sep 24 15:13 data drwxr-xr-x 3 spark spark 4096 Sep 24 15:13 ec2 drwxr-xr-x 3 spark spark 4096 Sep 24 15:13 examples drwxr-xr-x 2 spark spark 4096 Sep 24 15:13 lib -rw-r--r-- 1 spark spark 50972 Sep 24 15:13 LICENSE drwxr-xr-x 2 spark spark 4096 Nov 25 18:09 logs -rw-r--r-- 1 spark spark 22559 Sep 24 15:13 NOTICE drwxr-xr-x 6 spark spark 4096 Sep 24 15:13 python drwxr-xr-x 3 spark spark 4096 Sep 24 15:13 R -rw-r--r-- 1 spark spark 3593 Sep 24 15:13 README.md -rw-r--r-- 1 spark spark 120 Sep 24 15:13 RELEASE drwxr-xr-x 2 spark spark 4096 Oct 27 15:02 sbin drwxr-xr-x 2 spark spark 4096 Nov 25 14:07 work |
上記を/usr/local以下にsparkをディレクトリを作成しそこへコピーします。
|
1 2 |
mkdir /usr/local/spark cp -a * /usr/local/spark |
次にワーカーノードのホストの設定をします。
/usr/local/spark/confに移動し、slaves.templateをslavesとしてコピーします。
そして中身を「slavesの中身」のように編集します。
|
1 2 3 |
cd /usr/local/spark/conf cp -a slaves.template slaves vim slaves |
|
1 2 3 4 |
node1 node2 node3 node4 |
(上記までの作業はすべてのワーカーノード上でも実施する必要があります)
slavesファイルの編集が完了したら/usr/local/spark/sbinに移動し、Apache Sparkスタンドアローンクラスタのマスターとワーカーをsparkユーザーで起動します。
|
1 2 |
cd /usr/local/spark/sbin sudo -uspark ./start-all.sh |
上記実行後に各サーバーでps aux を実行するとマスターノードには
org.apache.spark.deploy.master.Master
各ワーカーノードには
org.apache.spark.deploy.worker.Worker
が起動しているがわかると思います。
HadoopではCDHを使用しない場合でも最低nodemanger,secondarynodemanger,datanode,resourcemanager,nodemanagerの起動が必要となり、CDHの場合にはより多くのデーモンの起動が必要になります。それに比べ、Apache Sparkは2デーモンのみなのでシンプルであることがわかると思います。
2.wikipedia記事データの変換とMongoDBへの登録
まず前提としてwp2txtはインストール済み、wikipedia記事アーカイブファイル(jawiki-latest-pages-articles.xml.bz2)はマスターノードにダウンロード済みのものとします。
以下のようにしてjawiki-latest-pages-articles.xml.bz2をテキストに変換します。
|
1 |
wp2txt --input-file jawiki-latest-pages-articles.xml.bz2 |
上記はbzip2形式のwikipedia記事アーカイブを解凍し、さらにXMLベースのデータを解析してテキストに変換してファイルとして出力してくれます。
出力されるファイルは1つではなく分割されて出力されます。(1ファイルにつき3000件程度の記事データが含まれています)
アーカイブにはおおよそ1000000記事分のデータが含まれており、すべての記事データをテキスト化するのを待つととても時間がかかります。
今回使用したのはリソースとの兼ね合いで100000件程度なので解凍・テキスト化途中で100000件分くらいテキスト化が終わった時点で処理を中断してください。
これでwikipedia記事アーカイブファイルの解凍とテキスト化が終わったら次はMongoDBに登録します。
pythonで以下のようなソースコード書き、wiki_insert_to_mongo.pyの名前で保存します。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 |
#-*- coding:utf8 -*- import sys reload(sys) sys.setdefaultencoding("utf8") from pymongo import * import traceback import re try: #mongosへ接続 m = MongoClient("mongodb://master:27018") #sparktestという名のDBを選択(生成) db = m.sparktest #wiki_texts_littleという名のコレクションを用意 coll = db.wiki_texts_little #すでにデータが登録されていたら削除 coll.remove({}) #sparktestのシャーディングを有効にする try: m.admin.command("enableSharding" , "sparktest") except Exception , inst: pass #シャーディング対象のコレクションとしてsparktest.wiki_texts_little設定し、シャーディングキーとしてtitlelenを設定 try: m.admin.command("shardCollection" , "sparktest.wiki_texts_little" , key={"titlelen":1}) except Exception , inst: pass for i in range(1 , 400): #wikipedia記事データテキストファイルを読み込む fo = open("/data/jawiki-latest-pages-articles.xml-" + str(i) , "r+") pt1 = re.compile(r'^\[\[.+\]\]\n$') pt2 = re.compile(r'[\|\*#\=\{\}\[\]\n\t]+') page_contents = {} ctitle = "" for line in fo: line = line.replace(" "," ") #タイトル判定 if pt1.match(line) is not None: #タイトルからwikipedia編集用特殊気記号を削除 ctitle = pt2.sub("",line) if page_contents.has_key(ctitle) == False: #タイトルごとに各記事データを1行ずつリストに追加するための準備 page_contents.setdefault(ctitle , []) else: #wikipedia記事データ(1行ごと)からwikipedia編集用特殊記号を削除してタイトル毎に用意されたリストに追加 page_contents[ctitle].append(pt2.sub("",line)) insert_data = [] #タイトル毎の記事データリストを読み込む for title , contents in page_contents.iteritems(): #MongoDBバルクインサート用リストに辞書型に整形した記事データを追加(記事データリストを全角空白で結合) insert_data.append({"title":title , "contents":" ".join(contents) , "titlelen":len(title)}) #MongoDBへ記事データをバルクインサート coll.insert_many(insert_data) except Exception , inst: print traceback.format_exc() |
保存したら実行します。多分2~3分程度で完了するかと思います。
|
1 |
python wiki_insert_to_mongo.py |
正常に登録されているか一応確認します。
|
1 2 3 |
mongo --host [mongosのIP] --port 27018 mongos>use sparktest mongos>show collections |
show collections実行後に表示されるコレクション一覧にwiki_texts_littleが存在しているかを確認します。
また100000万件登録されているかをコレクションのcount()メソッドで確認します。
|
1 |
mongos>db.wiki_texts_little.count() |
上記を実行して100000(多少オーバーでも想定の範囲内)と表示されていればOKです。
ついでに指定のカラムに指定のデータが登録されていることも確認しておきましょう。
|
1 |
mongos>db.wiki_texts_little.find().limit(1) |
limit指定をして1件だけ取得しましょう。
以下のような感じで登録されていればOKです。
|
1 |
{ "_id" : ObjectId("56556606eb0a196d9a4ec1ee"), "titlelen" : 12, "contents" : " 純粋数学(じゅんすいすうがく、pure mathematics)とは、しばしば応用数学と対になる概念として、応用をあまり意識しない数学の分野に対して用いられる総称である。 数学のどの分野が純粋数学でありどの分野が応用数学であるかという社会的に広く受け入れられた厳密な合意があるわけではなく、区別は便宜的なものとして用いられることが多い。また数学がより広範な範囲で利用されるに従い、分野としての純粋と応用との区別はあいまいで困難なものとなってきている。ただし、純粋数学という用語を用いる場合の志向としては、議論される数学の厳密性、抽象性をもととした数学単体での美しさを重視する傾向がある。 関連項目 応用数学 数理科学 ", "title" : "純粋数学" } |
ついでのついでに(最後です)コレクションwiki_texts_littleがシャーディングされていることを確認しておきます。
sh.status()を実行します。sh.から始まるコマンドはシャーディング関連のコマンドです。
|
1 |
mongos>sh.status() |
以下のように表示されたらOKです。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 |
--- Sharding Status --- sharding version: { "_id" : 1, "minCompatibleVersion" : 5, "currentVersion" : 6, "clusterId" : ObjectId("55f685ef2fc9ee37f7c339f5") } shards: { "_id" : "repl_1", "host" : "repl_1/[レプリカセット1プライマリのIP]:27017,[レプリカセット1セカンダリのIP]:27017" } { "_id" : "repl_2", "host" : "repl_2/[レプリカセット2プライマリのIP]:27017,[レプリカセット2セカンダリのIP]:27017" } balancer: Currently enabled: yes Currently running: no Failed balancer rounds in last 5 attempts: 0 Migration Results for the last 24 hours: No recent migrations databases: { "_id" : "admin", "partitioned" : false, "primary" : "config" } { "_id" : "sparktest", "partitioned" : true, "primary" : "repl_2" } sparktest.wiki_texts_little <-これ shard key: { "titlelen" : 1 } chunks: repl_1 12 repl_2 13 too many chunks to print, use verbose if you want to force print |
これでwikipedia記事データのMongoDBへの登録が完了しました。
3.mongo-hadoopコネクターのダウンロードとビルド
SparkアプリケーションからMongoDBへアクセスするためにmongo-hadoopコネクターのダウンロードとビルドを行います。
mongo-hadoopコネクターはgithubで管理されておりmongodb/mongo-hadoopからマスターブランチをダウンロードし、解凍します。
|
1 2 |
wget https://github.com/mongodb/mongo-hadoop/archive/master.zip unzip master.zip |
解凍後にmongo-hadoop-masterというディレクトリが生成されますのでその中へ移動し、ビルドします。
|
1 2 |
cd mongo-hadoop-master ./gradlew jar |
ビルド処理は依存ライブラリのダウンロードも含む関係上結構時間がかかります。
ビルド完了後、後述するsbtのライブラリディレクトリに移動してください。
|
1 |
cp -a core/build/libs/mongo-hadoop-core-1.5.0-SNAPSHOT.jar /usr/local/sbt/bin/lib |
4.sbtのインストールと設定
scalaを用いてSparkアプリケーションを開発するためにsbtのインストールと設定を行います。
sbtはsimple build toolの略称でJava、scalaアプリケーションの開発・ビルドを支援するものです。
sbtをhttp://www.scala-sbt.org/download.htmlからwgetでマスターノードにダウンロード後、解凍します。
|
1 2 |
wget https://dl.bintray.com/sbt/native-packages/sbt/0.13.9/sbt-0.13.9.tgz tar xfvz sbt-0.13.9.tgz |
sbtというディレクトリが生成されるのでその中身を/usr/local/sbtというディレクトリを作成してコピーして、/usr/local/sbt/bin以下にproject、src、libディレクトリを作成します。またsrc以下に更にmain/scalaディレクトリを作成します。
|
1 2 3 4 5 6 7 |
cd sbt mkdir /usr/local/sbt cp -a * /usr/local/sbt cd /usr/local/sbt/bin mkdir project mkdir -p src/main/scala mkdir lib |
projectディレクトリにはプロジェクト設定をscala形式で記述したbuild.scalaファイルと使用するsbtプラグインの情報を記述するplugins.sbtを配置します。
src/main/scalaディレクトリにはアプリケーションのソースを配置します。
次にプロジェクトの設定と使用するsbtプラグインの設定をします。
/usr/local/sbt/bin/projectに移動し、「編集後build.scala」の内容でbuild.scalaというファイル名で保存します。
|
1 2 |
cd /usr/local/sbt/bin/project vim build.scala |
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 |
import sbt._ import Keys._ object sparksamplesbuild extends Build { val Organization = "spark.samples" //パッケージ名 val Name = "sparksamples" //プロジェクト名 val Version = "0.0.1" //プロジェクトバージョン val ScalaVersion = "2.10.5" lazy val project = Project ( "sparksamples", //プロジェクト名? file("."), settings = Seq( organization := Organization, name := Name, version := Version, resolvers += Classpaths.typesafeReleases, javaOptions += "-Djava.library.path=/usr/local/lib", libraryDependencies ++= Seq( "org.apache.spark" % "spark-core_2.10" % "1.5.1" % "provided", //Apache Sparkコアライブラリ "org.apache.spark" % "spark-mllib_2.10" % "1.5.1" % "provided" //Spark MLlibライブラリ ) ) ) } |
build.scalaと同じディレクトリに「編集後plugins.sbt」の内容でplugins.sbtというファイル名で保存します。
|
1 |
vim plugins.sbt |
|
1 |
addSbtPlugin("com.eed3si9n" % "sbt-assembly" % "0.13.0") |
sbt-assemblyプラグインはsbtでjarファイルを生成できるようにするためのプラグインです。
特段指定しなくても開発したJava,scalaアプリケーション本体及び必要なライブラリが含まれるjarファイルをまとめて一つのjarファイルを生成してくれます。
次にsbtを起動します。
|
1 2 |
cd /usr/local/sbt/bin ./sbt |
上記実行後sbtのコマンドシェルが起動し、build.scalaの「libraryDependencies」に指定されたライブラリとplugins.sbtに指定されたプラグインをダウンロードされます。
最後に必要なサードパーティーライブラリのjarをsbtのライブラリディレクトに移動します。
/usr/local/sbt/bin/にlibとうディレクトリを作成してそこにサードパーティーライブラリのjarファイルを放り込んでおけばsbtで開発したアプリケーションビルド時に自動的に参照してくれます。build.scalaの「libraryDependencies」に使用したいライブラリを記述してもいいですがリポジトリにないライブラリに関しては手動でダウンロードとビルドを行い、このライブラリディレクトリに放り込む必要があります。
「0.下準備」でビルドしたmecab-JavaバインディングのMeCab.jarと「3.mongo-hadoopコネクターのダウンロードとビルド」でビルドしたmongo-hadoopコネクターのコアライブラリjarファイル(mongo-hadoop-core-1.5.0-SNAPSHOT.jar)を/usr/local/sbt/bin/libに移動してください。
これでやっとSparkアプリケーションを開発する準備が整いました。
5.wikipedia記事データクラスタリングSparkアプリケーションの開発とビルド
/usr/local/sbt/bin/src/main/scalaに移動して「sample.scala」の内容をsample.scalaのファイル名で保存します。
|
1 |
cd /usr/local/sbt/bin/src/main/scala |
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 118 119 120 121 122 123 124 125 126 127 128 129 130 131 132 133 134 135 136 137 138 139 140 141 142 143 144 145 146 147 148 149 150 151 152 153 154 155 156 157 158 159 160 161 162 163 164 165 166 167 168 169 170 |
package spark.samples import org.apache.hadoop.conf.Configuration import org.apache.spark.SparkConf import org.apache.spark.SparkContext import org.apache.spark.SparkContext._ import org.bson.BSONObject import org.bson.BasicBSONObject import com.mongodb.hadoop.{ MongoInputFormat, MongoOutputFormat, BSONFileInputFormat, BSONFileOutputFormat} import org.bson.types.ObjectId import scala.collection.JavaConversions._ import org.chasen.mecab.Tagger import org.chasen.mecab.Node import org.apache.spark.mllib.feature.HashingTF import org.apache.spark.mllib.clustering.{KMeans, KMeansModel} import org.apache.spark.mllib.feature.Normalizer import org.apache.spark.AccumulableParam object Sample { def main(args:Array[String]) = { println("this is sample.") try{ //MongoDB読み込み用設定 val mongoConfig = new Configuration() //MongoDBからの読み込み用DSN mongoConfig.set("mongo.input.uri" , "mongodb://master:27018/sparktest.wiki_texts_little") //Curosrからの読み込みのタイムアウトをオフ mongoConfig.set("mongo.input.notimeout" , "true") //spark 実行設定 val sparkConf = new SparkConf().setMaster("spark://master:7077").setAppName("spark sample") //シリアライザーをKryoに変更 sparkConf.set("spark.serializer", "org.apache.spark.serializer.KryoSerializer") //Kryoシリアライザーの最大バッファサイズ sparkConf.set("spark.kryoserializer.buffer.max" , "204800k") //akkaでの通信処理のスレッド数 sparkConf.set("spark.akka.threads" , "5") //RDDの圧縮を有効化 sparkConf.set("spark.rdd.compress" , "true") //Sparkアプリケーションの実行コンテキストクラスインスタンス生成 val sc = new SparkContext(sparkConf) //wikipedia記事データをMongoDBから読み込むのための準備 val docs = sc.newAPIHadoopRDD( mongoConfig,//MongoDB読み込み設定 classOf[MongoInputFormat],//入力データ型 classOf[Object],//入力データのkeyのデータ型 classOf[BSONObject]//入力データのvalueのデータ型 ) println("checkpoint1") //MongoDBからwikipedia記事データを読み込み(page_wordsは以下wiki単語リストと呼ぶ) val page_words = docs.map { case (k , v) => { //mecab使用準備 val mecab = new Tagger("-Ochasen") //wikipedia記事データの記事本文を形態素解析して改行コードで分割 val parsed = mecab.parse(v.get("contents").toString).split("\n").flatMap { p => { //特定品詞の単語のみ抽出 if (p == "EOS" || p == "") None else { val cols = p.split("\t") val word = cols(0) val features = cols(3).split("-") val feature = features(0) val sub_feature = if (features.size > 1) features(1) else "" if (feature == "名詞" && (sub_feature == "一般" || sub_feature == "サ変接続" || sub_feature == "固有名詞")) { Some(word) } else None } } } //wikipedia記事データのタイトルと、記事本文から抽出した単語リストをタプルにセット Tuple2(v.get("title").toString , parsed) } //単語リスト内の単語数が1以上のデータのみ抽出 } filter{ case(k,v) => v.size > 0 } //各Executorにwiki単語リストをキャッシュ page_words.cache() println("checkpoint2") //wiki単語Mapを生成 val word_map = sc.accumulable(scala.collection.mutable.Map.empty[String,Int])(WordMap) page_words.foreach { case (title , words) => { words.map { word => { word_map += word } } } } val input = page_words println("checkpoint3") //wiki単語リストの単語の整数ID化と特徴量算出を実施後org.apache.spark.mllib.linalg.Vector型にして最後に特徴量の正規化を実施(wordvecsを以下wiki単語ベクターと呼ぶ) val tf = new HashingTF(word_map.value.keys.toList.size) val normalizer1 = new Normalizer() val wordsvecs = input.map { case (title , words) => { normalizer1.transform(tf.transform(words)) } } //上記で生成したwiki単語ベクターを各Executor毎にキャッシュ wordsvecs.cache() println("checkpoint4") //k-meansでwikiベクターをクラスタリングする準備(10グループにクラスタリング) val clusters = KMeans.train(wordsvecs , 10 , 20) println("checkpoint5") //上記で作成したk-meansのモデルを使ってwiki単語リストをクラスタリングしてその結果をMongoDBに格納するために整形する(ouputを以下wikiクラスタリング後データと呼ぶ) val output = input.map { case (title , words) => { //記事毎にwiki単語ベクターを生成 val wordsvec = tf.transform(words) //上記で作成したk-meansモデルでwiki単語ベクターを評価 val cluster_idx = clusters.predict(wordsvec) //Bsonオブジェクトを生成してカラムpage_titleに記事タイトル、cluster_idxにクラスタIDを設定 val bson = new BasicBSONObject() bson.put("cluster_idx" ,cluster_idx.toString) bson.put("page_title" , title) Tuple2(new ObjectId() , bson) } } println("checkpoint6") //MongoDBへの書き込み設定 val outputConfig = new Configuration() outputConfig.set("mongo.output.uri","mongodb://master:27018/sparktest.clustered_result") //MongoDBにwikiクラスタリング後データを書き込む output.saveAsNewAPIHadoopFile( "file:///this-is-completely-unused",//未使用なので""でもいい classOf[Object],//出力データのkeyのデータ型(MongoDBの_idに相当) classOf[BSONObject],//出力データのvalueのデータ型 classOf[MongoOutputFormat[Object, BSONObject]],//出力データ型 outputConfig) } catch { case e:Exception => e.printStackTrace() } 0//これは戻り値ですが適当です } } //カスタムアキュムレーターの定義 object WordMap extends AccumulableParam[scala.collection.mutable.Map[String,Int] , String] { def zero(map1:scala.collection.mutable.Map[String,Int]) = scala.collection.mutable.Map.empty[String,Int] //WordMapをマージ def addInPlace(map1:scala.collection.mutable.Map[String,Int] , map2:scala.collection.mutable.Map[String,Int]) = { map2.foreach { case (k,v) => { if (map1.contains(k) == false) map1.put(k , v) } } map1 } //WordMapに単語を追加 def addAccumulator(map1:scala.collection.mutable.Map[String,Int] , val1:String) = { if (map1.contains(val1) == false) { map1.put(val1 , 1) } map1 } } |
次にコンパイルですが、その前にソースコードを簡単に説明します。
(コメントの通りの箇所は割愛させていただきます)
|
1 2 3 4 5 6 7 8 9 10 11 12 |
//spark 実行設定 val sparkConf = new SparkConf().setMaster("spark://master:7077").setAppName("spark sample") //シリアライザーをKryoに変更 sparkConf.set("spark.serializer", "org.apache.spark.serializer.KryoSerializer") //Kryoシリアライザーの最大バッファサイズ sparkConf.set("spark.kryoserializer.buffer.max" , "204800k") //akkaでの通信処理のスレッド数 sparkConf.set("spark.akka.threads" , "5") //RDDの圧縮を有効化 sparkConf.set("spark.rdd.compress" , "true") //Sparkアプリケーションの実行コンテキストクラスインスタンス生成 val sc = new SparkContext(sparkConf) |
上記はSparkアプリケーションの実行用設定を行っている部分です。
setMasterではApache Sparkスタンドアローンクラスターのマスターの設定「spark://[スタンドアローンクラスターのマスタ]:[使用ポート]」の形式で設定をしてます。
setMasterを使用する代わりに後述するspark-submitの引数としてマスター情報を渡すことも可能です。
setAppNameでこのSparkアプリケーションの名前を設定してます。これはなんでもOKです。
後はSparkアプリケーション実行時に参照されるいくつかの設定の変更をしています。
spark.serializerにはSparkアプリケーション内で扱うデータを別のサーバーに送信する場合のシリアライズ用クラスを指定します。デフォルトはjava.io.Serializerですが今回はテキストデータを別サーバーに送信することになるためちょっとでも送信するデータ量を減らしておきたいと思い、より小さいサイズに圧縮できるKryoシリアライザーを使用しています。このシリアライザーは特別な準備をする必要なく標準で使用することができます。
spark.kryoserializer.buffer.maxは詳細はわかりませんがシリアライズ時のバッファの最大サイズを指定するものです。今回わざわざ明示的に設定してあるのはデフォルトのままだとバッファが足りないというエラーメッセージを出力し例外を投げて異常終了してしまうためです。
後の設定はコメントのまんまです。早くなるかもとなんとなく設定してあるのでどこまでSparkアプケーションの実行スピードに影響を及ぼすかは不明です。
最後にSparkアプリケーション実行用設定クラスインスタンスを引数として実行コンテキストインスタンスを生成してます。この実行コンテキストがSparkアプリケーションの実行を制御する役割を担っているため必須となります。
|
1 2 3 4 5 6 7 |
//wikipedia記事データをMongoDBから読み込むのための準備 val docs = sc.newAPIHadoopRDD( mongoConfig,//MongoDB読み込み設定 classOf[MongoInputFormat],//入力データ型 classOf[Object],//入力データのkeyのデータ型 classOf[BSONObject]//入力データのvalueのデータ型 ) |
sc.newAPIHadoopRDDは本来HDFSからSparkアプリケーション用入力データを読み込むためのものですがmongo-hadoopコネクターを導入し入力データ型を変更することによりMongoDBからのデータの読み込みが可能となります。戻り値はRDD[(K,V)]となります。ちなみにRDDは正直私も完全な理解を得られていませんが、透過的に分散処理されるコレクションだと考えておけばいいでしょう。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 |
//MongoDBからwikipedia記事データを読み込み(page_wordsは以下wiki単語リストと呼ぶ) val page_words = docs.map { case (k , v) => { //mecab使用準備 val mecab = new Tagger("-Ochasen") //wikipedia記事データの記事本文を形態素解析して改行コードで分割 val parsed = mecab.parse(v.get("contents").toString).split("\n").flatMap { p => { //特定品詞の単語のみ抽出 if (p == "EOS" || p == "") None else { val cols = p.split("\t") val word = cols(0) val features = cols(3).split("-") val feature = features(0) val sub_feature = if (features.size > 1) features(1) else "" if (feature == "名詞" && (sub_feature == "一般" || sub_feature == "サ変接続" || sub_feature == "固有名詞")) { Some(word) } else None } } } //wikipedia記事データのタイトルと、記事本文から抽出した単語リストをタプルにセット Tuple2(v.get("title").toString , parsed) } //wiki単語リスト内の単語数が1以上のデータのみ抽出 } filter{ case(k,v) => v.size > 0 } |
上記は基本的にコメントの通りですがcase (k , v)とTuple2(v.get(“title”).toString , parsed)の箇所のみ説明します。
case (k , v)は想像つく方もいらっしゃるかもしれませんがkはMongoDBから読み込んだデータの_id、vはMongoDBから読み込んだデータ(BSON)となります。
Tuple2(v.get(“title”).toString , parsed)はタプル要素の1番目にwikipedia記事のタイトルを、2番目は記事本文を形態素解析した後の単語リストとなります。
この形式をmapの戻り値とすると暗黙的にRDD[(K,V)]の型のRDDが生成され、読み込み時にcase (k , v)という読み出し方が可能となるためこの形式にしてあります。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 |
//wiki単語Mapを生成 val word_map = sc.accumulable(scala.collection.mutable.Map.empty[String,Int])(WordMap) page_words.foreach { case (title , words) => { words.map { word => { word_map += word } } } } ....... //カスタムアキュムレーターの定義 object WordMap extends AccumulableParam[scala.collection.mutable.Map[String,Int] , String] { def zero(map1:scala.collection.mutable.Map[String,Int]) = scala.collection.mutable.Map.empty[String,Int] //各Executor毎のWordMapをマージ def addInPlace(map1:scala.collection.mutable.Map[String,Int] , map2:scala.collection.mutable.Map[String,Int]) = { map2.foreach { case (k,v) => { if (map1.contains(k) == false) map1.put(k , v) } } map1 } //WordMapにない単語を追加 def addAccumulator(map1:scala.collection.mutable.Map[String,Int] , val1:String) = { if (map1.contains(val1) == false) { map1.put(val1 , 1) } map1 } } |
上記は全wiki単語リストを走査してユニークなwiki単語リスト(厳密にはMapですが)を生成してます。
これは実のところ必要がないのですが、Sparkアプリケーション内で使用できるアキュムレーターと言われる分散環境共有変数の説明と単語リストの単語の整数IDへの変換と特長量算出のために使用するHashingTFのコンストラクタの引数として重複を除いた語彙数を渡すために用意したロジックです。語彙数を渡さなくても動作しますが、その場合には自動的に2の20乗(1048576)となります。
まずアキュムレーターですがこれはApache Spark分散環境下で共有され、かつ値を加算することのみが可能な変数です。標準のアキュムレーターは基本的に整数型、浮動小数点型などの基本データ型のみが用意されており、主に集計に使用されます。
但し、今回使用しているものは標準のアキュムレーターではなく、カスタムアキュムレーターと呼ばれるものです。
これはAccumulableParamを継承して定義された開発者独自のアキュムレータを指します。上記のWordMapを見るとわかりますが、定義されている3つのzero,addInPlace,addAccumulatorをオーバーライドして標準サポート以外のデータ型にも対応したアキュムレーターを定義することになります。
3つのメソッドのそれぞれの働きはzeroはアキュムレータの初期化、addInPlaceが各Executor毎のアキュムレーターの統合、addAcumulatorがアキュムレータへの値の追加となります。
WordMapのようにカスタムアキュムレータを定義したのち、
|
1 |
val word_map = sc.accumulable([初期値])(カスタムアキュムレータ名) |
と言った形で初期化することにより使用することができるようになります。
|
1 2 |
//k-meansでwikiベクターをクラスタリングする準備(10グループにクラスタリング) val clusters = KMeans.train(wordsvecs , 10 , 20) |
上記はk-meansでwiki記事データをクラスタリングするため、Spark MLlibのKMeansオブジェクトのtrainメソッドを学習用データ、クラスタ数、各クラスタ毎の中心の再計算の回数を引数に呼び出しています。trainメソッドはクラスタ数と再計算の回数が多くなればなるほど負荷が高くなります。今回100000件のデータをクラスタするにあたって少ないように見える10を指定したのは用意したインフラリソースではこれが限界であるためです。(10以上の値も試しましたがOut of Memoryでこけるし、今回は事例を挙げるのが目的なので穏便に完了するようクラスタ数を10としました)
|
1 2 3 4 5 6 7 8 9 10 11 12 13 |
val output = input.map { case (title , words) => { //記事毎にwiki単語ベクターを生成 val wordsvec = tf.transform(words) //上記で作成したk-meansモデルでwiki単語ベクターを評価 val cluster_idx = clusters.predict(wordsvec) //Bsonオブジェクトを生成してカラムpage_titleに記事タイトル、cluster_idxにクラスタIDを設定 val bson = new BasicBSONObject() bson.put("cluster_idx" ,cluster_idx.toString) bson.put("page_title" , title) Tuple2(new ObjectId() , bson) } } |
上記はwiki単語リストからMongoDBへの出力データを生成しています。
wiki単語リストをwiki単語ベクターに変換し、それを学習後のk-meansのモデルクラスであるKMeansModelクラスインスタンスのpredictメソッドに渡してwiki単語ベクターの属するクラスタの判定を行っています。その後、MongoDBへの出力準備としてデータ形式をBSONオブジェクトに変換しています。
ここでmapの戻り値がTuple2(new ObjectId() , bson)となっていますが、これも前出のTuple2(v.get(“title”).toString , parsed)と同じで、この形式を戻り値とすると暗黙的にRDD[(K,V)]に変換されて返却されるためです。
但し、最後の
|
1 2 3 4 5 6 7 |
//MongoDBにwikiクラスタリング後データを書き込む output.saveAsNewAPIHadoopFile( "file:///this-is-completely-unused",//未使用なので""でもいい classOf[Object],//出力データのkeyのデータ型(MongoDBの_idに相当) classOf[BSONObject],//出力データのvalueのデータ型 classOf[MongoOutputFormat[Object, BSONObject]],//出力データ型 outputConfig) |
で呼び出しているsaveAsNewApiHadoopFileはRDD[(K,V)]のメソッドではなくorg.apache.spark.rdd.PairRDDFunctionsというクラスのメソッドです。
saveAsNewApiHadoopFileメソッド呼び出し時にRDD[(K,V)]が暗黙的にorg.apache.spark.rdd.PairRDDFunctionsクラスのインスタンスに変換されるようです。
逆に言うと出力用のデータの形式をTuple2(K,V)にしておかないと、RDD[(K,V)]に変換されず、しいてはsaveAsNewApiHadoopFileを呼び出せないということになります。というかコンパイル時にエラーになります。(Javaではこの暗黙変換は適用されません。明示的な変換が必要になります。)
ちなみにsaveAsNewAPIHadoopFileは本来HadoopのHDFSへSparkアプリケーションでの処理結果を出力するためのメソッドですが、入力データ読み込み時と同じく引数をMongoDBへの出力用に設定してやるとMongoDBへ出力することができます。
ではソースコードの説明はここまでとしてコンパイルを実施します。
|
1 2 |
cd /usr/local/sbt/bin ./sbt compile |
コンパイルがエラーなく完了したら次は必要なライブラリをひとまとめにしたjarファイルを生成します。
|
1 |
./sbt assembly |
上記を実行後、/usr/local/sbt/bin/target/scala-2.10以下にsparksamples-assembly-0.0.1.jarが生成されます。
これでSparkアプリケーションの開発とビルドは完了です。
6.Sparkアプリケーションの実行
それでは開発したSparkアプリケーションを実行してみましょう。
ますはSparkアプリケーションの本体が含まれるjarファイルをマスターノードとワーカーノードの同じ場所にコピーします。後述するspark-submit実行時にコピー場所を指定すると自動的にその場所から所定の場所(DriverとExecutorが起動されたワーカーノードの/usr/local/spark/work以下)にコピーされます。
「5.Sparkアプリケーションの開発とビルド」で生成されたsparksamples-assembly-0.0.1.jarを/var/tmpにコピーします。
|
1 2 |
cd /usr/local/sbt/bin/ cp -a target/scala-2.10/sparksamples-assembly-0.0.1.jar /var/tmp |
各ワーカーノードへのコピーは今回rsyncを使用しました。以下のような感じで各ワーカーノードへコピーします。
|
1 2 |
cd /var/tmp sudo -uspark rsync -auvzr -e ssh sparksamples-assembly-0.0.1.jar /var/tmp |
それではよいよ実行に移ります。
/usr/local/spark/binに移動し、spark-submitコマンドを実行し、Sparkアプリケーションを起動します。
|
1 2 3 4 5 6 7 8 9 10 11 |
cd /usr/local/spark/bin sudo -uspark ./spark-submit --supervise \ --driver-memory 4G \ --total-executor-cores 4 \ --executor-memory 4G \ --executor-cores 1 \ --verbose \ --master spark://master:7077 \ --deploy-mode "cluster" \ #スタンドアローンクラスタ環境でSparkアプリケーションを起動 --class spark.samples.Sample5 \ /var/tmp/sparksamples-assembly-0.0.1.jar |
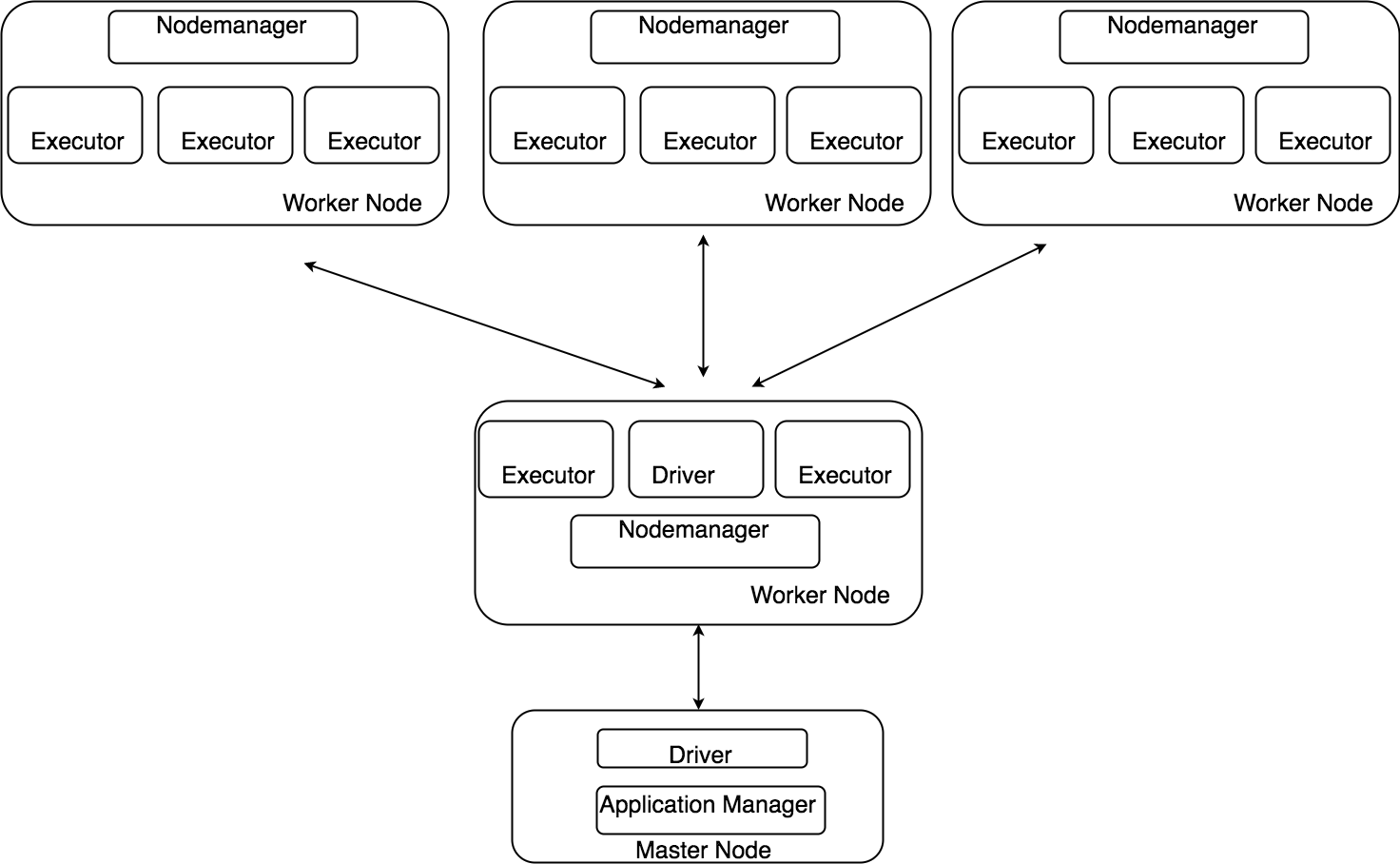
spark-submitはDriverとExecutorを起動し、–class引数で渡されたクラスに含まれるSparkアプリケーションを起動します(Driverが起動してるのかspark-submitが起動しているのかは詳細は調べてません)。Driverは起動されたSparkアプリケーションの実行制御(分散対象となる処理をExecutorに渡す等)と分散対象となっていない処理の実行、Executorは分散対象となっている処理(RDDのmapメソッドの中の処理等)を実行します。
Driver、Executor共に/usr/local/spark/conf/slavesに記述されたワーカーノードのいずれかで起動されます。Executorは全てのワーカーノードで起動され、ドライバーは自動選択されたワーカーノード1台に1つだけ起動されます。Driverもそこそこメモリを必要とするため、起動するノードの選択ができないか調べましたがどうも現時点ではできないようです。(spark.driver.hostというSparkアプリケーションの実行時設定が存在するがこれを設定してもエラーになる)
引数の説明を簡単にしますと
–driver-memory:Driverの使用可能メモリ上限
–total-executor-cores:起動されるExecutorが同時に使用可能なCPUコア数上限
–executor-memory:Executor毎の使用可能メモリ上限
–executor-cores:Executor毎の使用可能なCPUコア数の上限
–master:マスター情報(今回はSparkアプリケーションの内部で設定しているので多分不要ですが一応設定しました)
–deploy-mode:Sparkアプリケーションの実行環境の指定(スタンドアローン環境・スタンドアローンクラスタ環境・Hadoop-YARN環境等)
と言った感じになります。
–superviseはSparkアプリケーションのエラーハンドリングの厳密化と耐障害性のアップのためのものかなとは思いますが、厳密な意味は不明です。今回は何となく指定してあります。
基本的にそこそこ重要なもののみしか指定してませんが、–total-executor-coresは特に重要です。ここの数値を単純にワーカーノードの台数x各ワーカーノードのCPUコア数としてしまうと各ワーカーノードに場合によっては限界以上にExecutorが起動されてしまう場合があります。何の限界かというとCPUではなく、メモリです。CPUのパワーはあまり使わないがExecutor毎メモリをかなり必要とするSparkアプリケーションを実行する場合、これは小さめにしておく方がいいです。今回はこれを知らずに大き目に設定したところ限界を超えて(100%中の180%くらい)メモリを使用しようとしてSparkアプリケーションが落ちてしまいました。
spark-submit実行後以下のような感じでメッセージが表示されれば起動は成功です。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 |
Using properties file: null Parsed arguments: master spark://master:7077 deployMode cluster executorMemory 4G executorCores 1 totalExecutorCores 4 propertiesFile null driverMemory 4G driverCores null driverExtraClassPath null driverExtraLibraryPath null driverExtraJavaOptions null supervise true queue null numExecutors null files null pyFiles null archives null mainClass spark.samples.Sample primaryResource file:/var/tmp/sparksamples-assembly-0.0.1.jar name spark.samples.Sample childArgs [] jars null packages null packagesExclusions null repositories null verbose true Spark properties used, including those specified through --conf and those from the properties file null: spark.driver.memory -> 4G Running Spark using the REST application submission protocol. Main class: org.apache.spark.deploy.rest.RestSubmissionClient Arguments: file:/var/tmp/sparksamples-assembly-0.0.1.jar spark.samples.Sample System properties: spark.driver.memory -> 4G spark.executor.memory -> 4G spark.cores.max -> 4 SPARK_SUBMIT -> true spark.driver.supervise -> true spark.app.name -> spark.samples.Sample spark.jars -> file:/var/tmp/sparksamples-assembly-0.0.1.jar spark.submit.deployMode -> cluster spark.master -> spark://master:7077 spark.executor.cores -> 1 Classpath elements: Using Spark's default log4j profile: org/apache/spark/log4j-defaults.properties 15/11/26 14:54:37 INFO RestSubmissionClient: Submitting a request to launch an application in spark://master:7077. 15/11/26 14:54:37 WARN RestSubmissionClient: Unable to connect to server spark://master:7077. Warning: Master endpoint spark://master:7077 was not a REST server. Falling back to legacy submission gateway instead. Main class: org.apache.spark.deploy.Client Arguments: --supervise --memory 4G launch spark://master:7077 file:/var/tmp/sparksamples-assembly-0.0.1.jar spark.samples.Sample System properties: spark.driver.memory -> 4G spark.executor.memory -> 4G spark.cores.max -> 4 SPARK_SUBMIT -> true spark.driver.supervise -> true spark.app.name -> spark.samples.Sample spark.jars -> file:/var/tmp/sparksamples-assembly-0.0.1.jar spark.submit.deployMode -> cluster spark.master -> spark://master:7077 spark.executor.cores -> 1 Classpath elements: |
Driverでの処理の経過はDriverが起動されたワーカーノードの/usr/local/spark/work/work-[JOBID]以下のstderrファイル、Executorでの処理の経過はワーカーノードの/usr/local/spark/work/app-[JOBID]以下のstderrファイルで確認することができます。
また、ソースコード中のprintlnの内容はすべてDriverのstderrファイルに出力されるようになっています。
7.結果の確認
完了したかどうかはログ(Driverの処理経過ログ)で確認することができます。
標準でWeb経由で処理経過を確認できるツールが付属しており(スタンドアローンクラスタマスター起動時に同時に自動的に起動されます)そこから確認できますが、今回は割愛します。
正常にwikipedia記事データのクラスタリング結果がMongoDBのclustered_resultに格納されているか確認します。
|
1 2 3 |
mongo --host [mongosのIP] --port 27018 mongos>use sparktest mongos>show collections |
show collections実行後に表示されるコレクション一覧にclustered_resultが存在することを確認します。
また件数・内容も確認します。
|
1 2 |
db.clustered_result.count() db.clustered_result.find() |
count()メソッド実行後に100000以上の数値が表示されればOkです。
また中身について、実際の出力内容は以下のようになります。(内容は抜粋です。下記のようなデータが実際には100000件あります)
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 |
{ "_id" : ObjectId("5655a095644415489fca3875"), "cluster_idx" : "8", "page_title" : "パサールカード" } { "_id" : ObjectId("5655a094644415489fca3842"), "cluster_idx" : "8", "page_title" : "熊本市電田崎線" } { "_id" : ObjectId("5655a093644415489fca383c"), "cluster_idx" : "8", "page_title" : "京急ウィング号" } { "_id" : ObjectId("5655a094644415489fca3847"), "cluster_idx" : "8", "page_title" : "熊本市電健軍線" } { "_id" : ObjectId("5655a094644415489fca3854"), "cluster_idx" : "8", "page_title" : "北関東自動車道" } { "_id" : ObjectId("5655a094644415489fca3864"), "cluster_idx" : "8", "page_title" : "発電設備の運用" } { "_id" : ObjectId("5655a094644415489fca386d"), "cluster_idx" : "8", "page_title" : "泉北高速鉄道線" } { "_id" : ObjectId("5655a094644415489fca386e"), "cluster_idx" : "8", "page_title" : "北陸鉄道石川線" } { "_id" : ObjectId("5655a095644415489fca3873"), "cluster_idx" : "8", "page_title" : "新幹線700系電車" } { "_id" : ObjectId("5655a08f644415489fca375c"), "cluster_idx" : "8", "page_title" : "総合車両製作所" } { "_id" : ObjectId("5655a08e644415489fca3746"), "cluster_idx" : "8", "page_title" : "小田急小田原線" } { "_id" : ObjectId("5655a090644415489fca378e"), "cluster_idx" : "8", "page_title" : "シルバーゴール" } { "_id" : ObjectId("5655a091644415489fca37bc"), "cluster_idx" : "8", "page_title" : "埼玉高速鉄道線" } { "_id" : ObjectId("5655a092644415489fca37eb"), "cluster_idx" : "8", "page_title" : "ホームライナー" } { "_id" : ObjectId("5655a093644415489fca3821"), "cluster_idx" : "8", "page_title" : "エアポート快特" } { "_id" : ObjectId("5655a093644415489fca382a"), "cluster_idx" : "8", "page_title" : "新交通システム" } { "_id" : ObjectId("5655a093644415489fca3830"), "cluster_idx" : "8", "page_title" : "原子間力顕微鏡" } { "_id" : ObjectId("5655a093644415489fca3832"), "cluster_idx" : "8", "page_title" : "放射線・環状線" } { "_id" : ObjectId("5655a079644415489fca3342"), "cluster_idx" : "8", "page_title" : "弘南鉄道大鰐線" } { "_id" : ObjectId("5655a079644415489fca3348"), "cluster_idx" : "8", "page_title" : "弘南鉄道弘南線" } |
なんとなーく似た者同士が同じクラスタに振り分けられていることが確認できるかと思います。
最後に
こんな感じでApache Sparkを使うと簡単にビッグデータを入力とした機械学習処理(当然集計でもOK)ができることが分かってもらえたかと思います。
スタンドアローンクラスター環境を試す場合にはどうしても複数台のサーバーが必要になるため個人でためすにはちょっと敷居が高いかもしれませんがConoHa等でなら低コストで試すことも可能かと思いますので是非一度Apache Sparkに触れてみてください。